9. 更多有用模块
本章节包含诸多快捷的模块方便配合EasyMultiProfiler数据分析流程使用。
9.1 多重字符串匹配检测 str_detect_multi
注意:
①模块str_detect_multi可以同时匹配多个感兴趣的字符串。
②参数
①模块str_detect_multi可以同时匹配多个感兴趣的字符串。
②参数
exact支持模糊查找和完全匹配查找。text <- c('Bacilli_unclassfiled','Bacteroidia_uncuture','Other')
str_detect_multi(text,c('Bacilli','bacteroidia'),exact=FALSE) # Ignore the capital letter
str_detect_multi(text,c('Bacilli','Bacteroidia'),exact=TRUE) # Set the matched completely
此外,在EasyMultiProfiler数据分析流程中,模块str_detect_multi也可以提供帮助。
🏷️示例1:可以同时提取微生物组学中门级别中Bacteroidetes和Firmicutes的物种。
MAE |>
EMP_assay_extract('taxonomy') |>
EMP_filter(feature_condition = str_detect_multi(Phylum,c('Bacteroidetes','Firmicutes')))
🏷️示例2:可以剔除在纲级别没有完整注释的物种。
MAE |>
EMP_assay_extract('taxonomy') |>
EMP_filter(feature_condition = !str_detect_multi(Class,'unclassified'))
9.2 兼容EasyMicroPlot数据分析流程 EMP_to_EMP1
在EasyMultiProfiler数据分析流程中,可以利用函数将微生物数据快速导出成EasyMicroPlot可接受的格式。
注意:
①模块EMP_to_EMP1导出数据时需要首先利用EMP_feature_convert将微生物注释切换为全注释。
②模块EMP_to_EMP1无法导出已经折叠完毕的数据。
①模块EMP_to_EMP1导出数据时需要首先利用EMP_feature_convert将微生物注释切换为全注释。
②模块EMP_to_EMP1无法导出已经折叠完毕的数据。
🏷️示例:导出微生物数据,并完成EasyMicroPlot的共发生网络分析。
# Get the data from EasyMultiProfiler
MAE |>
EMP_assay_extract('taxonomy') |>
EMP_feature_convert(from = 'tax_single',add = 'tax_full') |>
EMP_to_EMP1(estimate_group = 'Group') -> deposit
# Work in the EasyMicroPlot
library(EasyMicroPlot)
cooc_re <- cooc_plot(data = deposit$data,design = deposit$mapping,
meta = deposit$meta,min_relative = 0.001,
min_ratio = 0.7,cooc_method'spearman',
cooc_output = TRUE)
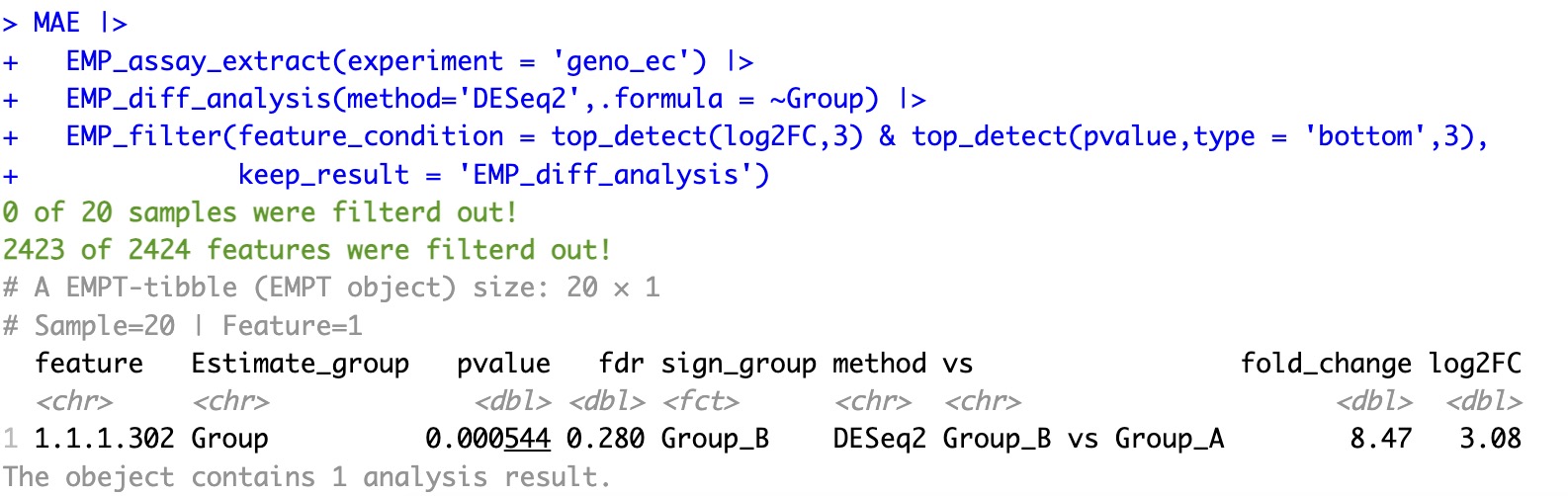
9.3 最大或者最小数值检测 top_detect
注意:
当输入的n为一个0到1的小数时,则按照百分比提取。
当输入的n为一个0到1的小数时,则按照百分比提取。
这个函数可以帮助EasyMultiProfiler数据分析流程快速筛选出想要的最高或者最低的数据。
🏷️示例: 选出差异分析中,log2FC最大的三个特征与pvalue最小的三个特征的交集。
MAE |>
EMP_assay_extract(experiment = 'geno_ec') |>
EMP_diff_analysis(method='DESeq2',.formula = ~Group) |>
EMP_filter(feature_condition = top_detect(log2FC,3) & top_detect(pvalue,type = 'bottom',3) ,
keep_result = 'EMP_diff_analysis')
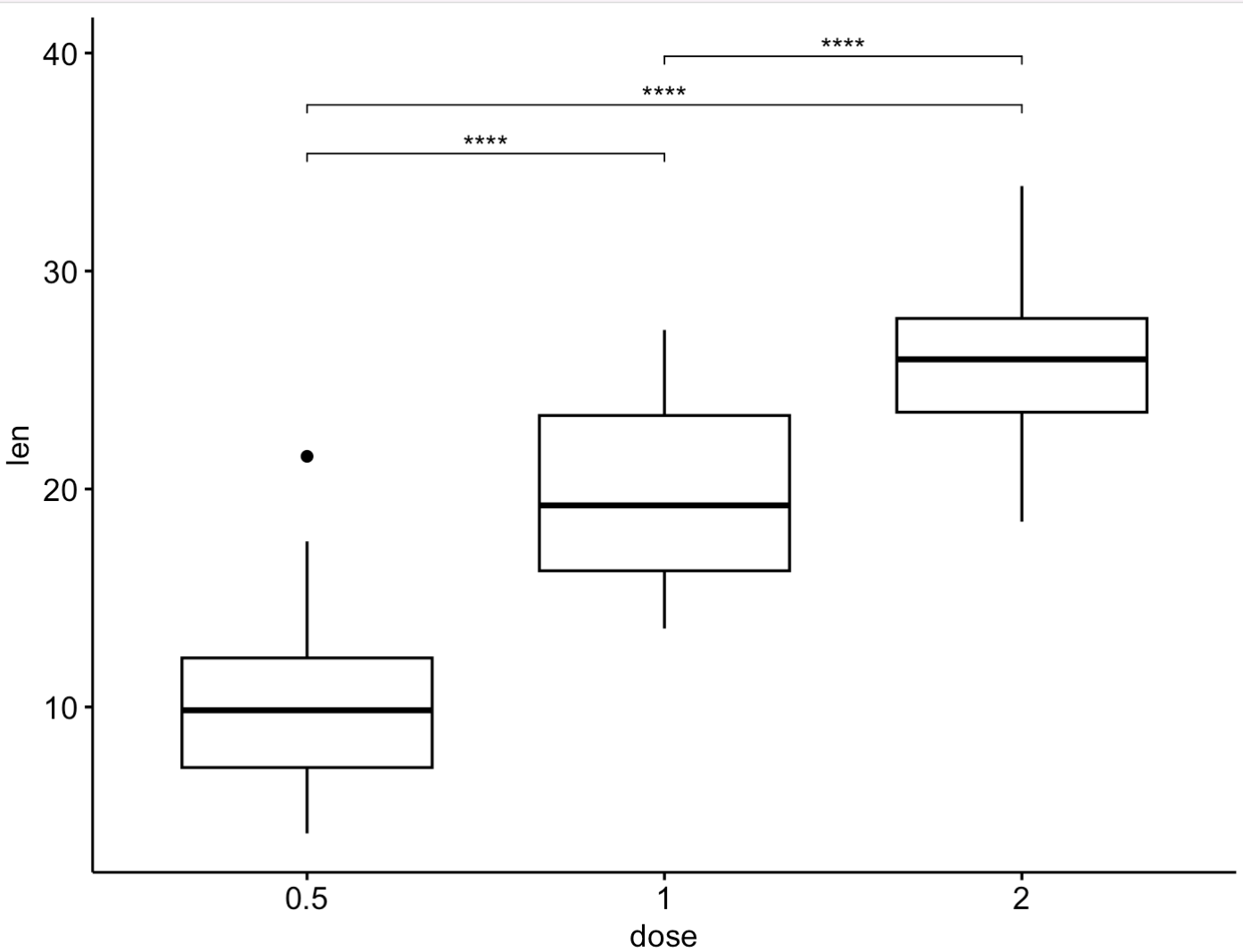
9.4 多组差异分析 stat_test
此函数能够针对表格,快速实现多组件的差异分析比较,并自动设置合适的位置辅助ggplot2绘图。
🏷️示例1: 常规比较
data(ToothGrowth)
# 指定分组
stat_test(
data = ToothGrowth,
estimate_group = 'dose',
value = 'len',
method = "t.test"
)
# 使用公式
stat_test(
data = ToothGrowth,
formula = len ~ dose,
method = "t.test"
)
🏷️示例2: 亚组比较
data(ToothGrowth)
# 指定分组
stat_test(
data = ToothGrowth,
value = 'len',
estimate_group = "supp",
compare_group = "dose",
method = "t.test"
)
# 使用公式
stat_test(
data = ToothGrowth,
estimate_group = 'supp',
formula = len ~ dose,
method = "t.test"
)
🏷️示例3: 对接ggplot2图形
stat_result <- stat_test(
data = ToothGrowth,
formula = len ~ dose,
value = 'len',
method = "tukey.hsd",
)
# for ggpubr
if(require("ggpubr")){
ggboxplot(ToothGrowth, x = "dose", y = "len") +
stat_pvalue_manual(stat_result, label = "p.signif", tip.length = 0.01)
}